GitHubを使えって絶対に言うな!!!
えーっと、BCGワクチン接種が新型コロナウイルス感染症(COVID-19)に有効説検証論文に付随した話で、同一世界にワクチン接種ありとなしの集団がいたらどうなんの? — と言う御題があって、(ドラクエのマップのような縦横がつながっている)トーラス世界にマス目を区切って、そこに接種ありと接種なしの人々をランダムに配置して、隣接したマス目にいる人からのみ影響を受けるようなトイモデルで、どうなるか検討してみました。
大事なポイントなので強調しますが、以下のような感じで、乱雑にワクチン有りと無しを詰め込みます。
| 有 | 有 | 無 | 有 | 有 |
| 有 | 無 | 無 | 無 | 無 |
| 無 | 無 | 有 | 無 | 無 |
| 無 | 有 | 有 | 無 | 無 |
| 有 | 無 | 無 | 有 | 無 |
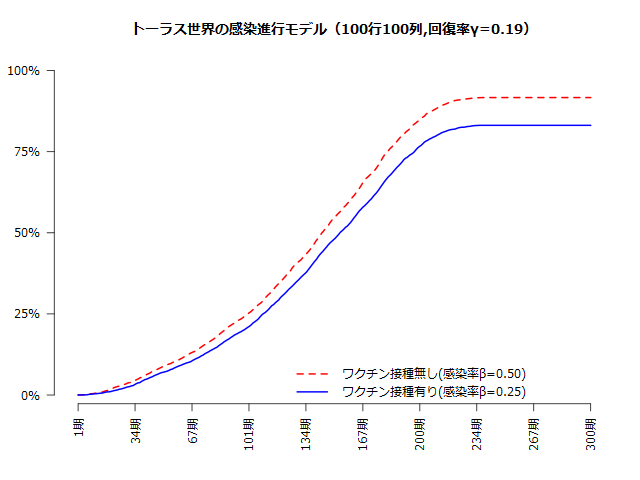
まぁ、ワクチンに効果があればワクチンを打つ方が感染しないですし、集団免疫獲得につれて効果量が特に落ちると言うわけでもないです。ただし、ワクチン有効率は最初が大きく中盤で落ち着きます。
しかし、詳しく特異なモデルをつくってもSIRモデルに近づくんですよね。やはり古典的な微分方程式モデルは強い。
set.seed(202004)
# 感染率β
beta_0 <- 0.50 # ワクチン非接種者
beta_1 <- 0.25 # ワクチン接種者# 回復率γ
gamma <- 0.19# ワクチン接種者の比率
theta <- 0.5# トーラス状の空間を表すn行n列の行列を複数つくる
n <- 100# 計算する期間
t <- 250# ワクチン接種者は1で、他は0とする
shot <- numeric(n^2)
shot[order(runif(n^2))[1:as.integer(n^2*theta)]] <- 1# ワクチン接種の有り無しの人数を数えておく
obs <- tapply(shot, shot, length)# ワクチン接種の有無を表す行列
m_shot <- matrix(shot, n)# 最初に感染しているのは1人
inf <- numeric(n^2)
inf[order(runif(n^2))[1]] <- 1# 感染者を表す行列
m_inf <- matrix(inf, n)# 暴露レベルを表す行列を返す
m_exposed <- function(m_inf){
# 感染者の左側を1とする行列(トーラスなのに注意)
inf <- c(m_inf)
m_l <- matrix(c(inf[-(1:n)], inf[1:n]), n)
# 感染者の右側を1とする行列
inf <- c(m_inf)
m_r <- matrix(c(inf[((n^2-n+1): n^2)], inf[-((n^2-n+1): n^2)]), n)
# 感染者の上を1とする行列
inf <- c(t(m_inf))
m_u <- matrix(c(inf[-(1:n)], inf[1:n]), n, byrow=TRUE)
# 感染者の下を1とする行列
inf <- c(t(m_inf))
m_b <- matrix(c(inf[((n^2-n+1): n^2)], inf[-((n^2-n+1): n^2)]), n, byrow=TRUE)# 0~4の値が返る
m_l + m_r + m_u + m_b
}# 完全な免疫を獲得した回復者を表す行列
m_rcv <- matrix(numeric(n^2), n)# データフレームを作成する
df1 <- data.frame(
t = 1:t,
pct_infected_shot = numeric(t),
pct_infected_no_shot = numeric(t),
number_of_infected = numeric(t),
VE = numeric(t)
)for(i in 1:t){
# 感染確率を表す行列
# = ウイルスに暴露されている×社会的距離×{世代に応じたβ}×非回復者
p <- (beta_0 - c(m_shot) * (beta_0 - beta_1)) * abs(c(m_rcv) - 1)
# 周囲にいる感染者の人数に応じて、感染確率が上がる
m_prob <- matrix(1 - (1 - p)^c(m_exposed(m_inf)), n)# 感染を判定し、感染者行列を更新
m_inf[runif(n^2) < c(m_prob)] <- 1
# 回復判定をし、感染者行列と免疫保持者行列を更新
flag <- (runif(n^2) < gamma)&(m_inf==1)
m_inf[flag] <- 0
m_rcv[flag] <- 1# 感染経験者は、現在感染者もしくは回復者
infected <- c(m_inf)|c(m_rcv)
# ワクチン接種者と非接種者ごとに感染経験者を集計
noi <- tapply(infected, c(m_shot), sum)
# ワクチン接種者と非接種者ごとに感染経験率を出す
pct <- noi/obs
df1$number_of_infected[i] <- sum(noi)
df1$pct_infected_no_shot[i] <- pct[1]
df1$pct_infected_shot[i] <- pct[2]
df1$VE[i] <- 1 - pct[2]/pct[1]
}title <- sprintf("トーラス世界の感染進行モデル(%d行%d列,回復率%.2f,接種率%.2f)", n, n, gamma, theta)
windowsFonts(Meiryo = windowsFont("Meiryo"))
par(family="Meiryo", mar=c(5, 4, 4, 2), mgp=c(2, 1, 0), bg="white")plot(df1$pct_infected_no_shot, col="red", type="l", lwd=2, axes=FALSE, xlab="", ylab="", lty=2, ylim=c(0, 1), main=title)
lines(df1$pct_infected_shot, col="blue", lwd=2)y_at <- seq(0, 1, 0.25)
y_labels <- sprintf("%d%%", 100*y_at)
y_space <- abs(par()$usr[1] - par()$usr[2])/25
axis(2, at=y_at, labels=y_labels, pos=- y_space, las=2)x_at <- seq(1, t, length.out=10)
x_labels <- sprintf("%.0f期", x_at)
x_space <- abs(par()$usr[3] - par()$usr[4])/40
axis(1, at=x_at, labels=x_labels, pos=-x_space, las=2)legend("bottomright", lty=c(2, 1), lwd=c(2, 2), pch=c(-1, -1), col=c("red", "blue"), legend=c(sprintf("ワクチン接種無し(感染率β=%.2f)", beta_0), sprintf("ワクチン接種有り(感染率β=%.2f)", beta_1)), bty="n", y.intersp=1.25)
dev.copy(png, sprintf("%sβ0=%.2f,β1=%.2f.png", title, beta_0, beta_1), width=640, height=480, type="cairo", bg="white")
dev.off()
t2 <- as.integer(t/3)
df2 <- df1[1:t2,]
title <- sprintf("トーラス世界の感染進行モデル(%d行%d列,回復率%.2f,接種率%.2f)", n, n, gamma, theta)windowsFonts(Meiryo = windowsFont("Meiryo"))
par(family="Meiryo", mar=c(5, 4, 4, 2), mgp=c(2, 1, 0), bg="white")ylim <- c(0, ceiling(max(df2$pct_infected_no_shot)*10)/10)
plot(df2$pct_infected_no_shot, col="red", type="l", lwd=2, axes=FALSE, xlab="", ylab="", lty=2, ylim=ylim, main=title)
lines(df2$pct_infected_shot, col="blue", lwd=2)y_at <- seq(0, ylim[2], length.out=5)
y_labels <- sprintf("%.0f%%", 100*y_at)
y_space <- abs(par()$usr[1] - par()$usr[2])/25
axis(2, at=y_at, labels=y_labels, pos=- y_space, las=2)x_at <- seq(1, t2, length.out=10)
x_labels <- sprintf("%.0f期", x_at)
x_space <- abs(par()$usr[3] - par()$usr[4])/40
axis(1, at=x_at, labels=x_labels, pos=-x_space, las=2)legend("topleft", lty=c(2, 1), lwd=c(2, 2), pch=c(-1, -1), col=c("red", "blue"), legend=c(sprintf("ワクチン接種無し(感染率β=%.2f)", beta_0), sprintf("ワクチン接種有り(感染率β=%.2f)", beta_1)), bty="n", y.intersp=1.25)
dev.copy(png, sprintf("%sβ0=%.2f,β1=%.2f,前半.png", title, beta_0, beta_1), width=640, height=480, type="cairo", bg="white")
dev.off()