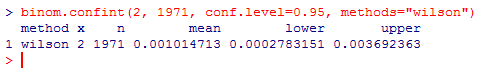
とあるJuliaを使った教材に、JuliaやNumPyのsumはSIMD拡張命令セットを使うから速いけれども、C言語でsumを書くとそうではないから遅いような記述があって、CからSIMDを叩けば良いだろうと思ったので、私にしては珍しくC99のコードで、単浮動小数点のsumを書いてみました。gccでしかコンパイルできません。
謳い文句によるとgccは自動的にSIMD拡張命令セットを使うことになっていますが、実際のところはsumのような単純なものでも使いません。こんなにSIMD向きの単純計算でも、128bitsを32bits×4で使っても2倍ちょっとしか速くならないわけで、倍精度だとAVX-256以降でないと意味がないからでしょうか。
なお、C++のテンプレート・ライブラリEigenは行列演算などにSIMD拡張命令セットを使ってくれるので、こういう苦しい記述をしないで浮動小数点演算で速度を出したい人はC++/Eigenを使うべきです。CPUのサポートしている命令セットごとにコードを書き換えないといけないって、手間隙な上に混乱してきますからね。
#include <stdio.h>
#include <stdlib.h>
#include <math.h>
#include <emmintrin.h>
#include <time.h>
#include <cpuid.h>float x87_sum(float *X, int n){
float s = 0.0;
for (size_t i = 0; i < n; ++i) {
s += X[i];
}
return s;
}float simd_sum(float *X, int n){
static int ws = sizeof(__m128)/sizeof(float);
__m128 r128, s128 = {0};
float s = 0.0, sws[ws];
int m = m % ws;// add by ws-float from [0 n-m]
for (size_t i = 0; i < n - m; i+=ws) {
r128 = _mm_load_ps(&X[i]);
s128 = _mm_add_ps(r128, s128);
}
// store result of the above code.
_mm_store_ps(sws, s128);// sum sws up, and record it into s.
for (size_t i = 0; i < ws; i++) {
s += sws[i];
}// add the rest, [n-m+1 n-1], by a float.
for (size_t i = 1; i <= m; i++) {
s += X[n - i];
}return s;
}int main(){
float *X;
int n = 10000;
int i;
long et;
int ws;
double r1, r2;
union cpuid_sig {
unsigned int i;
unsigned char c[4];
} sig;
unsigned int highest, eax, ebx, ecx, edx;highest = __get_cpuid_max(0x8000000, &sig.i);
if(0 < __get_cpuid(highest, &eax, &ebx, &ecx, &edx) && 0 != bit_SSE2 & edx){
printf("SSE2 is supported.\n");
} else {
fprintf(stderr, "SSE2 is NOT supported!\n");
exit(-1);
}ws = sizeof(__m128)/sizeof(float);
printf("sizeof(__m128)/sizeof(float) = %ld/%ld = %d (%ld)\n", sizeof(__m128), sizeof(float), ws, sizeof(__m128) - sizeof(float)*ws);// setup X
X = calloc(n, sizeof(float));
for(i=0; i<n; i++){
X[n] = sinf(0.314F*i);
}et = clock();
r1 = x87_sum(X, n);
printf("elapsed time of x87 sum: %f\n", (double)(clock() - et)/CLOCKS_PER_SEC);et = clock();
r2 = simd_sum(X, n);
printf("elapsed time of SSE2 sum: %f\n", (double)(clock() - et)/CLOCKS_PER_SEC);printf("the difference of results: %f\n", fabs(r1 - r2));
return 0;
}