ここ10年間ぐらいで多重代入法を用いた欠損データ処理が一般化しましたが、多重代入法を使わなくても多重代入法と同様の結果が得ることもできます。Fully Bayesian Imputation Methodと呼ばれている手法ですが*1、最尤法による単一代入(imputation model)と回帰分析(analysis model)の同時推定するだけで、頻度主義的にも扱えるので試してみましょう*2。
データセット
Listwise手法ではバイアスが入るように、MARなデータセットを作成します。
gen_data <- function(n, mu = c(2, -3), S = matrix(c(2, -1.5, -1.5, 2), 2, 2)){ library(mvtnorm) X <- rmvnorm(n, mu, S) beta <- c(1, -1, 1) y <- cbind(1, X) %*% beta + rnorm(n) x <- X[,1] z <- X[,2] p <- 1/(1 + exp(-(4 + y))) mx <- x mx[runif(n) < p] <- NA data.frame(y, x, mx, z) } set.seed(1029) df01 <- gen_data(50)
yが従属変数、xとzが欠損値なしの説明変数です。cov(x, z)=-1.5≠0なので、zからx、xからzが推定できます。xに欠損値をつくったのがmxになり、今回、推定で用いるのはこれです。xはベンチマークとしてのみ使います。切片項、x、zの係数はそれぞれ1、-1、1になります。
データ整理
データフレームから、欠損値も入った行列X_aと、欠損値を除いた行列X_c、欠損値だけの行列X_mの三つを作成します。
frml <- y ~ mx + z df02 <- model.frame(frml, data = df01, na.action = na.pass) y <- model.response(df02) X_a <- model.matrix(frml, df02) X_c <- X_a[complete.cases(X_a), ] X_m <- X_a[!complete.cases(X_a), ]
推定
最尤法をかけるわけですが、
- 線形回帰のための最尤法の目的関数の中で、補定処理のための最尤法を行う入れ籠構造になっている
- 補定は多変量正規分布を仮定した最尤法(実は複数の欠損に対応)
- 線形回帰のパラメーターになるσとβの他、補定に用いるパラメーターμ(多変量正規分布の平均)を同時推定
- 補定に用いるΣ(多変量正規分布の共分散行列)は、μとデータから作成(concentrated loglikelihood function)
- 1行1行は補定せず、全部の行をまとめて補定している(→大きなデータセットには対応できない)
ことに注意してください。
objf <- function(p){ sigma <- p[1] beta <- p[2:4] mu <- p[5:6] X0 <- apply(X_c[, -1], 1, \(x) x - mu) # 切片項は補定しないので、除外する Sigma <- X0 %*% t(X0) / (ncol(X0) - 1) invSigma <- chol2inv(chol(Sigma)) init.p <- rep(0, sum(is.na(X_a))) r_optim <- optim(init.p, function(p){ X_a[is.na(X_a)] <- p sum(dmvnorm(X_a[, -1], mu, Sigma, log = TRUE)) }, function(p){ # グラディエントは無くても動く na <- is.na(X_m) index <- 1:length(X_m) X_m[na] <- p Y <- X_m[, -1] - rep(mu, each = nrow(X_m)) g <- t(-invSigma %*% t(Y)) g[index[na] - nrow(X_m)] # gは切片項を除外した行列からの計算結果なので、-nrow(X_m)で位置をあわせる }, method = "BFGS", control = list(fnscale = -1)) X_a[is.na(X_a)] <- r_optim$par r <- sum(dmvnorm(X_c[,2:3], mu, Sigma, log = TRUE)) r <- r + sum(dnorm(y - X_a %*% beta, sd = sigma, log = TRUE)) cat(sprintf("convergence: %d, loglikelihood: %f", r_optim$convergence, r), "\n") r } init_p <- c(1, rep(1, 3), apply(df02, 2, mean, na.rm = TRUE)[2:3]) r_mnorm <- optim(init_p, objf, method = "L-BFGS-B", lower = c(1e-1, rep(-Inf, 5)), control = list(fnscale = -1, maxit = 1000), hessian = TRUE)
そこそこ待たされます。
比較のための推定
OLSとmiceでも推定しておきます。
r_lm_c <- lm(y ~ x + z, df01) r_lm_m <- lm(y ~ mx + z, df01) library("mice") df_mice <- mice(df01[,-2], m = 20, maxit = 50, method = "pmm", seed = 2024) miced_lm <- with(df_mice, lm(y ~ mx + z)) r_mice <- pool(miced_lm)
推定結果
表にまとめます。
推定量の比較
m <- matrix(c(coef(r_lm_c), coef(r_lm_m), r_mnorm$par[2:4], summary(r_mice)[, 2]), 4, byrow = TRUE) rownames(m) <- c("OLS/no missing", "OLS/list-wise", "ML/imputed", "mice/imputed") colnames(m) <- sprintf("beta %d", (1:ncol(m))-1) print(m)

今回の手法(ML/imputed)が、欠損値のある行を削除するOLS/Listwise手法よりも良い推定値を出していることが分かります。
完全データのOLSとの差異
d <- t(sapply(2:nrow(m), \(i) m[i, ] - m[1, ])) rownames(d) <- rownames(m)[-1] print(d)
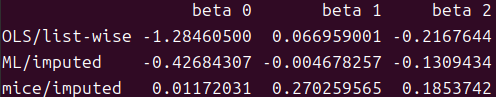
完全データの推定量との差分をとってmiceの結果と比較すると、切片項以外は今回の手法(ML/imputed)の方がmice推定量よりも完全データの推定量に近い推定量となっています。
標準誤差の比較
m.se <- matrix(c( coef(summary(r_lm_c))[,2], coef(summary(r_lm_m))[,2], sqrt(diag(solve(-r_mnorm$hessian)))[2:4], summary(r_mice)[,3] ), 4, byrow = TRUE) colnames(m.se) <- colnames(m) rownames(m.se) <- rownames(m) print(m.se)

単一代入だと標準誤差が過少推定される問題があるのですが、今回の手法(ML/imputed)の方がmice推定量よりも大きいので、過剰に帰無仮説を棄却する恐れは少ないことが分かります。
なぜ最尤法を使うのか?
多くの場合はmiceで十分間に合い、信頼のおけるパッケージと利用実績からmiceを使う方が望ましいのですが、この手法にも利点があります。ベイズ統計学への応用を考えると多重代入法は煩雑になりますが、尤度関数ですべて処理できれば簡単になります。また、補定処理に使うパラメーターも点推定されるので、実際に最尤推定量で代入された値が特定できるのは、プロットに使うときなどで便利なことが多いと思います。
なお、処理時間がかかりすぎるので使う気になれない場合は、補定処理を多変量正規分布の最尤推定から線形回帰の予測になどに書き換えれば時間短縮になります。複数の欠損値に対応できなくなりますが、実用上は特定列の欠損値を埋められれば足りることが多いでしょう。
*1:Best and Mason (2013) "Bayesian methods for missing data: part 1 — Key Concepts"を参考にしました。
*2:2年前に試しに書いたコード中でも使っていたりするのですが、今回は欠損値補定の部分をもっと一般化してみます。